SARS-CoV-2에 대한 새로운 통찰력을 제안하는 모델링 및 시뮬레이션
이전 블로그에서 우리는 잠재적인 약물 표적(예:단백질)의 초기 원자 수준 구조를 구축하고 실험적으로 결정할 수 없는 영역을 수정하기 위해 예측 모델링 도구를 사용하는 것에 대해 논의했습니다(동영상 참조 ). 이러한 도구에는 실험적으로 해결할 수 없는 수소 원자 및 유연한 루프의 추가가 포함됩니다. 우리는 최근 Science 저널에 발표된 SARS-CoV-2 스파이크(S) 단백질의 저온 전자 현미경(cryo-EM)의 맥락에서 이를 조사했습니다. (DOI:10.1126/science.abb2507).
이 블로그에서는 SARS-CoV-2 S 단백질과 같은 정제된 구조 모델의 분자 모델링 및 시뮬레이션이 COVID-19를 치료하기 위한 추정 치료제의 발견 및 설계를 위한 새로운 가설 생성에 어떻게 도움이 될 수 있는지 자세히 설명합니다.
약물 결합은 구조적 변화에 달려 있음
살아있는 시스템에서 단백질은 자연적으로 동적 개체로 존재합니다. 그들의 역학은 종종 그들의 기능을 미리 결정합니다. 물리학자 Richard Feynman은 다음과 같이 말했습니다.
“생명을 이해하려는 시도로 계속해서 이어지는 가장 강력한 가정을 들자면, 만물은 원자로 이루어져 있고 생물이 하는 모든 일은 다음과 같은 용어로 이해할 수 있다는 것입니다. 원자의 요동치는 소리."
1
SARS-CoV-2 S 단백질도 파인만의 원리에서 예외는 아닙니다. S 단백질은 인간 세포에 들어가기 전에 안지오텐신 전환 효소 2(ACE2)라고 하는 수용체에 결합합니다.
2
수용체 결합 도메인(RBD)은 ACE2에 결합하는 S 단백질의 일부입니다(그림 1A ). RBD는 up(수용기 접근 가능) 및 down(수용기 접근 불가) 상태라고 하는 최소한 두 가지 기본 형태 상태로 존재할 수 있습니다(그림 1B ). RBD가 up 상태에 있을 때 S 단백질은 ACE2의 결합을 촉진하기 위해 더 "개방"됩니다.
2
연구에 따르면 수용체에 접근할 수 없는 다운 상태가 더 안정적입니다.
2
이것은 RBD에 결합할 수 있는 작은 유기 분자와 같은 추정 치료제가 다운 상태에서 RBD를 안정화시키고 바이러스가 ACE2와 상호작용하는 것을 방지할 수 있음을 의미합니다. 따라서 COVID-19가 사람들을 감염시키는 것을 막습니다.
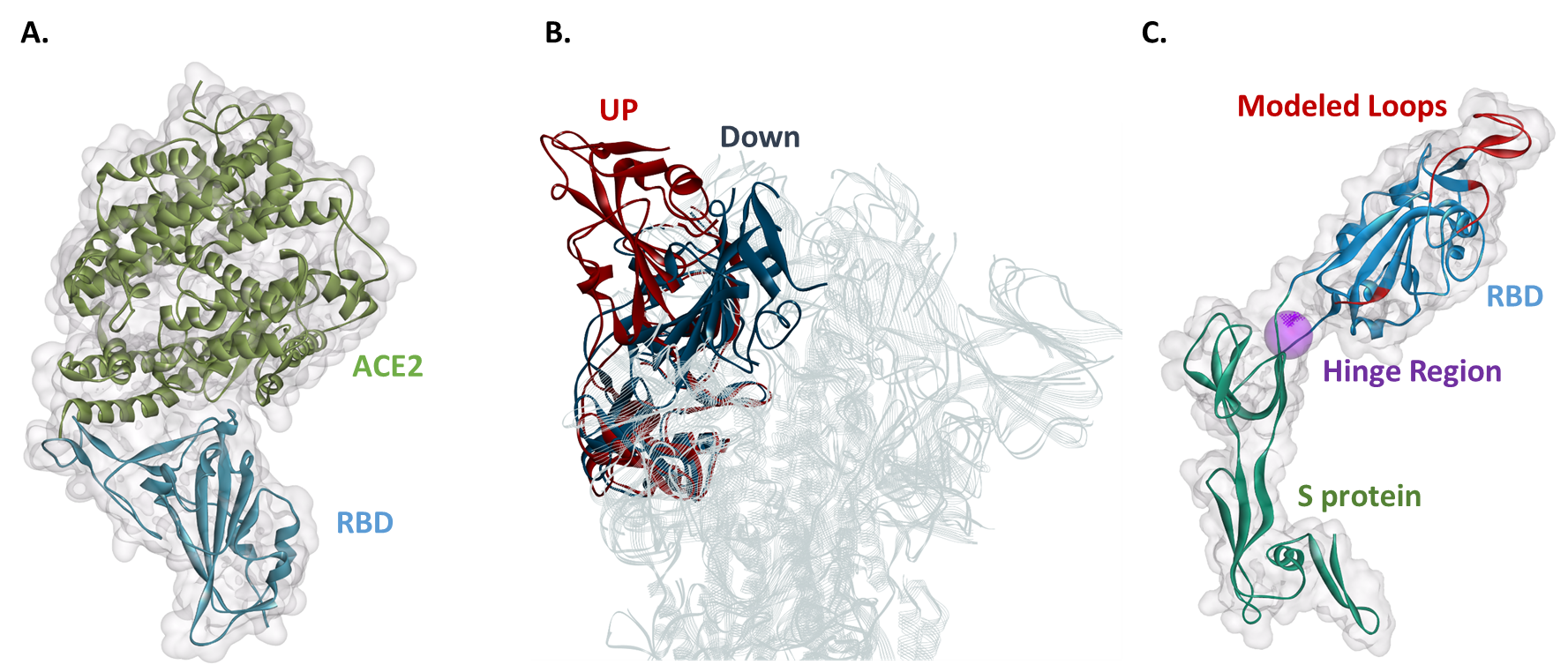
S 단백질의 RBD는 문에 달린 경첩과 같습니다.
유연한 링커는 RBD를 나머지 S 단백질에 연결합니다. 링커의 유연성으로 인해 RBD는 힌지 벤딩 동작을 통해 아래에서 위로 상태로 전환할 수 있습니다(그림 1B ). SARS-CoV-2 S 단백질에서 연결된 유연한 링커 및 인접 도메인이 있는 RBD를 추출했습니다(그림 1C ). 우리는 업 상태(PDB 6VYB)에서 S 단백질의 RBD 구조를 사용했지만 이 구조에는 ACE2를 결합하는 데 잠재적으로 중요한 세 개의 루프가 없습니다(그림 1C ). 결과적으로, 우리는 추가 템플릿과 함께 원래 절단이 만들어진 cryo-EM 구조(PDB 6VYB)를 사용하여 유연한 링커가 있는 RBD의 상동성 모델을 구축해야 했습니다. 추가 템플릿은 ACE2(PDB 6M17)와 복합된 RBD 단독의 결정 구조였습니다(그림 1A ).
RBD 구조는 열린 상태 구조(PDB 6VYB)에 존재하지 않는 20개 이상의 아미노산 잔기를 포함하는 루프를 포함합니다. 이 루프 중 두 개는 ACE2와의 상호 작용을 형성합니다. 따라서 Spike 단백질의 상호 작용을 이해하려면 상동성 모델링이 필요합니다. 그런 다음 pH와 같은 생리학적 조건을 모방하기 위해 적절한 양성자화 상태에서 수소 원자를 할당할 수 있습니다.
그런 다음 분자 역학(MD) 시뮬레이션을 수행하여 형태 전환을 시뮬레이션하고 추정되는 소분자가 결합하여 S 단백질과 ACE2의 상호 작용을 방해할 수 있는 가능한 결합 부위를 예측할 수 있습니다. 유연한 링커가 있는 RBD의 상동성 모델에 대해 BIOVIA Discovery Studio를 사용하여 가능한 결합 사이트를 예측했을 때 유연한 링커의 힌지에 위치한 결합 사이트를 식별했습니다(그림 1C ). 우리는 이 영역에 경첩이라는 레이블을 붙였으며 추정되는 치료법의 발견을 위해 추가 조사를 할 가치가 있을 수 있다는 점에 주목했습니다. 작은 분자가 힌지 영역에 결합하면 RBD를 다운 상태로 고정하여 ACE2 결합을 방지할 수 있습니다.
추가 조사
계산 예측 및 생물학적 실험을 포함한 광범위한 조사는 힌지 영역의 유용성을 더욱 명확히 할 수 있습니다. 계산 예측의 예에는 일반 모드 분석(NMA) 및/또는 MD 시뮬레이션이 포함될 수 있습니다.
3
예를 들어, MD 시뮬레이션의 긴 시간 척도(약 수백 나노초)를 통해 과학자들은 힌지 영역의 여러 형태를 샘플링할 수 있습니다.
반면에 NMA는 형태 전환에 대한 대략적이고 빠른 추정을 제공할 수 있습니다.
3
MD 시뮬레이션 및/또는 NMA의 특정 형태는 잠재적인 소분자 데이터베이스의 고처리량 가상 스크리닝을 위한 시작점입니다. 과학자들은 각 작은 분자를 도킹하고 모든 형태에 점수를 매길 수 있습니다. 그런 다음 모든 결과 포즈의 순위를 지정하고 실험적 검증을 위해 최고의 히트를 제출할 수 있습니다. 연구에 따르면 종종 앙상블 기반 가상 스크리닝이라고도 하는 컴퓨터 기반 약물 발견 방법이 약물 후보를 식별할 가능성을 높입니다.
4
이 방법은 또한 위에서 언급한 바와 같이 약물 결합이 단백질의 구조적 변화에 의존한다는 현실을 반영합니다. 여기에서 확인된 예비 결과는 흥미롭고 추가 조사할 가치가 있다고 생각합니다.
둘째, 정제된 S 단백질 구조 모델이 면역 치료제의 표적으로 사용될 수 있다는 점에 주목하고 싶습니다.
5
과학자들은 ACE2 결합 부위에 대한 사전 지식을 기반으로 SARS CoV-2 S 단백질에 결합하는 단일클론 항체를 설계할 수 있습니다. 그런 다음 실리코 공연을 할 수 있습니다. 결합 특이성을 개선하기 위한 친화성 성숙 연구.
6
BIOVIA Dassault Systèmes는 오늘날 COVID-19 솔루션에 대해 협력하고 있는 과학 커뮤니티의 적극적인 후원자로서 BIOVIA Discovery Studio를 개발합니다. 이 입증된 생명 과학 모델링 및 시뮬레이션 환경은 30년 이상의 동료 검토 연구와 세계적 수준의 실리코(in silico)를 결합합니다. 기법. 이 소프트웨어는 생물제제 설계 및 분석, 고전적 시뮬레이션, 구조 및 단편 기반 설계, 가상 리간드 스크리닝, ADME 및 독성 예측을 위한 도구를 포함하여 표적 식별에서 리드 최적화까지 사용할 수 있는 완전한 도구 세트를 과학자들에게 제공합니다.
Dassault Systèmes의 기업의 사회적 책임의 일환으로 BIOVIA는 SARS-CoV-2 관련 연구와 관련된 적격 학술 연구 그룹에 BIOVIA Discovery Studio에 대한 무료 6개월 라이선스를 제공하게 된 것을 기쁘게 생각합니다. SARS-CoV-2 바이러스에 대한 신속하고 안전하며 효과적인 치료제 후보를 찾는 데 도움이 됩니다. 이 분야의 학술 연구원이라면 소프트웨어 라이선스를 요청하고 다운로드하십시오. 이 제안은 2020년 6월 30일까지 진행됩니다.




